【好消息】16S测序项目之PICRUSt功能预测纳入“常规分析”!

为了将 16S 扩增测序的高性价比发挥到极致,建立起微生物基因组学与其他组学,尤其是代谢组研究的桥梁,阅微基因将把原本属于 16S 扩增子测序高级分析的 PICRUSt 功能预测归为常规分析,也就是说,还是一样的测序价格,您就能享受到原本需要额外加费用的高级分析。
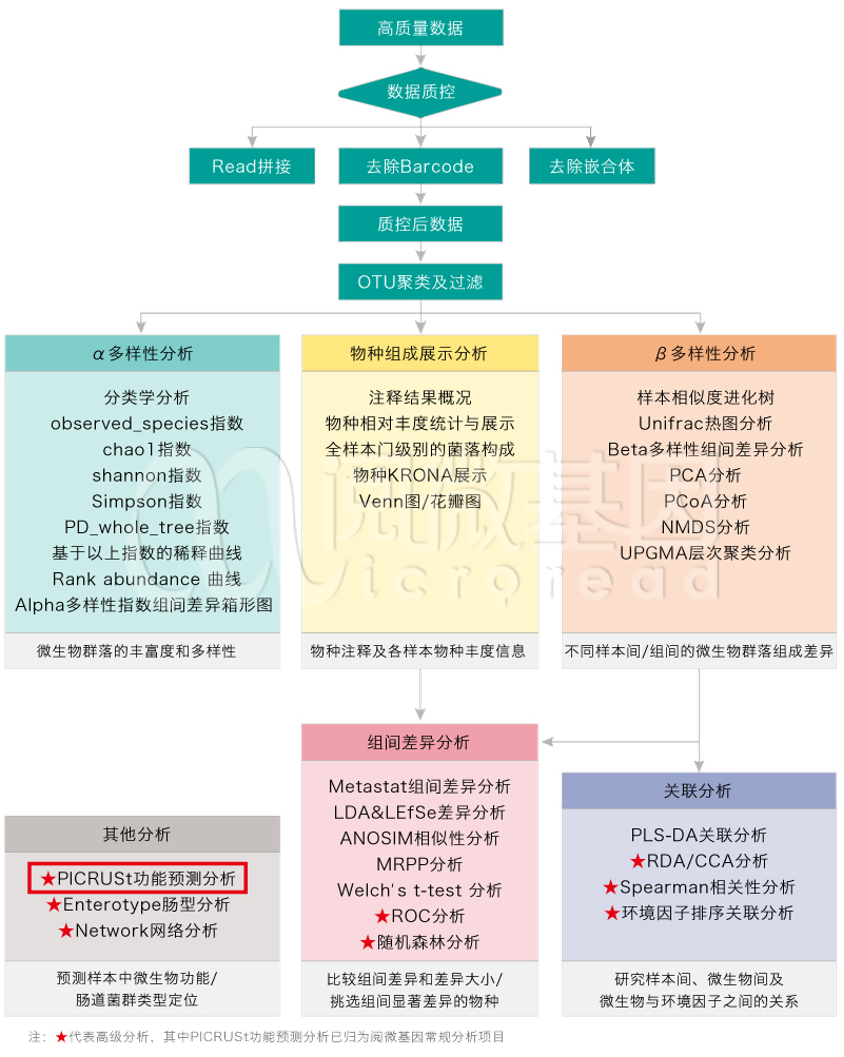

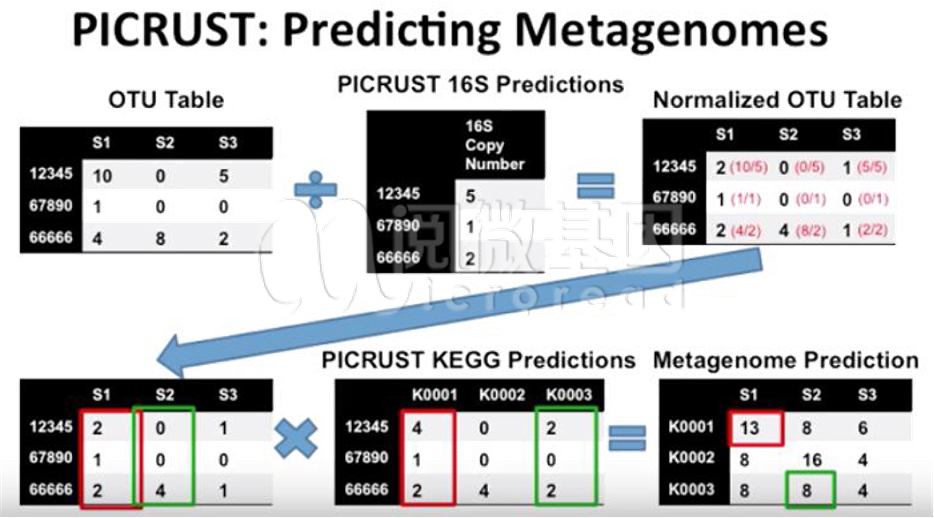
PICRUSt 工具全称是 Phylogenetic Investigation of Communities by Reconstruction of Unobserved States。此工具基于已测细菌 16S rDNA 的信息和对比 Greengene 数据库后近缘物种的 OTU 信息,推断它们的共同祖先的基因功能谱,同时对 Greengenes 数据库中其它未测物种的基因功能谱进行推断,构建古生菌和细菌域全谱系的基因功能预测谱;最后,将测序得到的菌群组成「映射」到数据库中,继而对菌群代谢功能进行预测。
2. 标准化 OTU 表格 (根据近缘物种的 16S rRNA 拷贝数进行校正);
3. 根据对应的近缘物种功能基因数 counts,结合预测标准化 OTUs counts 进行计算,最终得到样本和其功能基因的表格;
4. 对功能预测结果进行下一步分析,包括差异代谢通路分析、聚类热图分析、STAMP 分析等。

● 非常靠谱;PIRCUSt 功能预测的准确性达到 85%
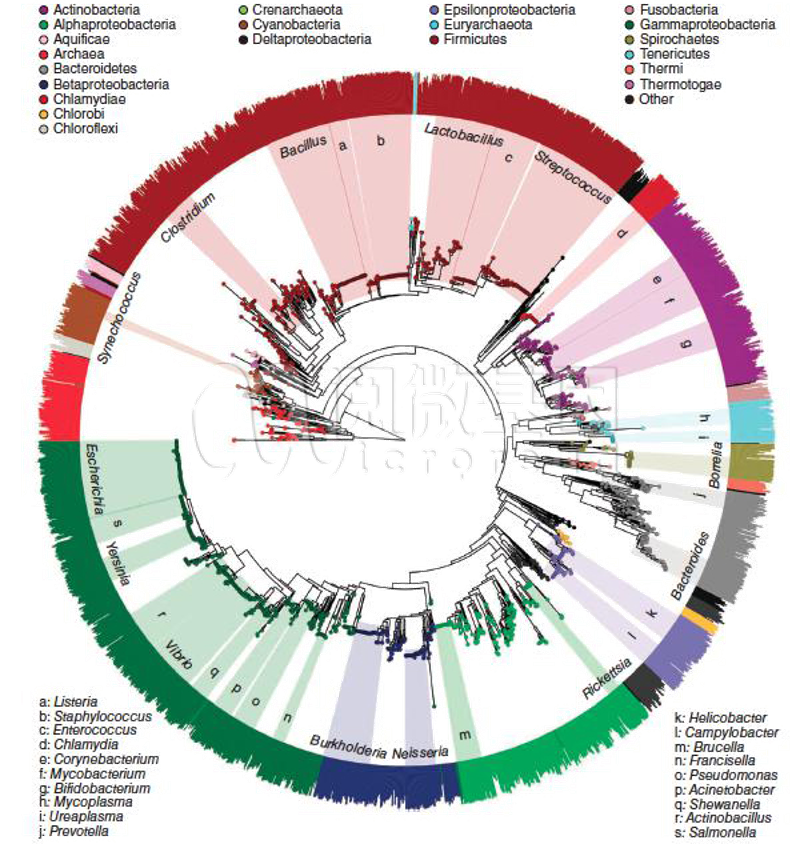
2013 年当 PICRUSt 算法发表时,开发者做了大量的研究和验证。令人欣喜的是,利用 PICRUSt 算法基于 16S rDNA 预测到的菌种基因组结果与真实的菌种参考基因组图谱非常接近,古菌预测准确度为 94%±4%,验证样本为 103 个;细菌预测准确度为 95%±5%,验证样本为 2487 个。如下图所示:
除了对 16S 基因组预测准确性的验证外,开发者对功能预测的准确性也做了验证,在不同的功能等级上,功能预测的准确性范围在 80%-100%,平均值达到 85%。

PICRUSt 虽然有着很好的预测准确率,但也存在着局限性。对于在 Greengenes 数据库里没有同源参考基因组序列的物种是无法被预测的;且只能对细菌和古生菌做功能预测,无法对真菌进行功能预测。
除此之外,PICRUSt 对于不同样本类型的微生物,其预测效果是不一样的。对人类或者哺乳动物的微生物预测效果最好, 对参考微生物基因组不全的样本类型,例如土壤和盐碱地,预测效果就会下降。

● 引用超多:PICRUSt 自 2013 年发表以来共被引用 1640 次,2018 一年超 600 次

PICRUSt 方法于 2013 年 9 月发表在《Nature Biotechnology 》杂志上,引用该文献的 SCI 文章也呈现逐年递增的趋势,总引用次数 1640 次,在刚刚过去的 2018 年,被引用次数高达 633 次,其中不乏《Cell 》、《Nature 》和《Science 》等顶级期刊。
● 除了 PICRUSt 之外,还有哪些功能预测的工具?
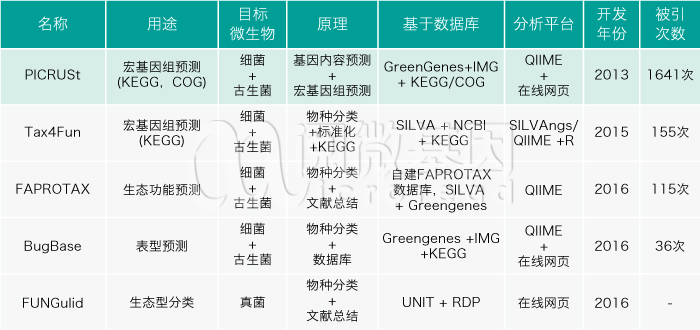
上表总结出了目前微生物领域的功能预测软件,PICRUSt 推出的最早,也有着最多的引用次数,最受科研人员认可和喜欢。但随着大家对古菌、真菌等多种非细菌群体的关注和注释数据库的更迭,也不断有其他新的算法推出。
参考文献:
[1] Langille MGI, Zaneveld J, Caporaso JG, McDonald D, Knights D, et al. (2013) Predictive functional profiling of microbial communities using 16S rRNA markergene sequences. Nature Biotechnology. DOI:10.1038/nbt.2676







